電気ビリビリごはんもぐもぐ
この記事は今年読んだ一番好きな論文Advent Calendar の一部として書かれました。
更新が停滞しがちな一年でしたが、論文は読んでいたのでこれなら記事かける!ウェイ!と思ってAdvent Calendar登録ページを見に行ったら初日しか空いてませんでした。トップバッターこわい。
というわけでこんなノリで大丈夫かしらと一抹の不安を抱えつつご紹介する「今年読んだ一番好きな論文」はこちらです。
とりあえず動画
まずは論文のSupplementary Videoを見てみましょう。
なんか知らんけどカッコよくない?ヤバくない?
よくわかんないけどヤバそう、どういうことなの
さきほどの動画は、枯草菌Baccilus subtilisのバイオフィルムを顕微鏡で観察し、細胞の負電荷を蛍光色素で可視化したものです。そこで、数mm規模の巨大な*1範囲に及ぶ振動現象が観察されました。
この論文で、微生物が電気信号を用いて情報伝達を行い、細胞同士で代謝状態を共有していることが初めて明らかになりました。
あたししょうがくせい。よくわかんない。
ぼくおなかすいたばいきん。ごはんたべたい。でんきビリビリしたら、ちかくのともだちがごはんゆずってくれたよ。うれしい。ごはんもぐもぐ。いきる。生きる。
ちゃんと論文解説しろ
内容の解説に入る前に、この論文ができるまでの背景をすこし説明したいと思います。
本論文を発表したカリフォルニア大学サンディエゴ校のGürol Süelラボでは、しばらくまえにBacillus subtilisのバイオフィルムの成長速度が振動する不思議現象を発見しました。
調べてみると、バイオフィルムが大きくなるにつれて細胞成長に必要な栄養素「グルタミン」がバイオフィルム内部で枯渇するため、その補充に使う時間を稼ぐためにバイオフィルム外縁部の細胞が成長を一時停止し、バイオフィルム内部の細胞を餓死させないように「配慮」してバイオフィルム全体の持続的な成長を可能にしているということがわかりました。
(この研究もNature論文になっていて面白いのでおすすめです。Süelラボによる解説動画もかわいいのでどうぞ。)
Metabolic co-dependence in bacterial biofilms
この成長振動論文の段階で振動現象の理由(Why)はわかったのですが、バイオフィルム全体で栄養状態の情報を共有して成長速度を調整するメカニズム(How)は不明なままでした。
そこで、筆者らはグルタミンの材料であるグルタミン酸とアンモニウムがそれぞれ荷電していることから、それぞれの代謝効率に細胞の電荷が関わっているのではないか?と仮説を立て、そこから長距離情報伝達法の候補としてイオンチャネルに目をつけました。
広く知られているように、動物の神経細胞はイオンチャネルを介して電気信号をやりとりすることで情報伝達を行っています。これまで微生物においてもイオンチャネルの存在自体は知られていましたが、実際の機能は謎に包まれていました。
筆者らは、まずはバイオフィルムの荷電ダイナミクスを可視化すべく、正電荷をもつ蛍光色素であるThioflavin-T(ThT)でバイオフィルムを処理し、独自のマイクロ流体デバイスで顕微鏡観察したところ、冒頭の動画に見られるような周期的な蛍光振動を発見しました。つまり、バイオフィルム全体で負電荷が伝播され、高度に同期していることが初めてわかったのです。そこでひとつ前の論文でみられた成長速度振動との関係を探ってみたところ、電荷振動と成長速度振動が見事に逆位相を示します。
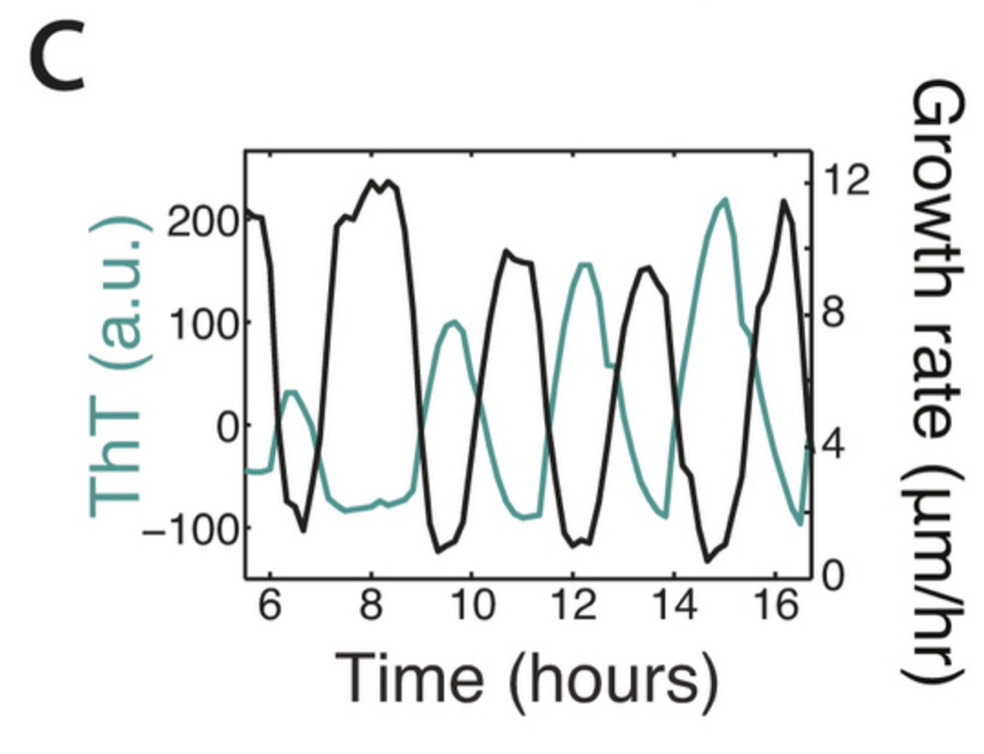
これはなにかあるぞ、ということで筆者らはいろいろ実験を重ね、
- グルタミン添加がThT振動を抑制すること*2
- 細胞外カリウム濃度がThTと同期して振動していること
- 細胞内カリウム濃度と同程度のカリウムをKClのかたちで添加するとThT振動が抑制されること
- 拡散では説明できない速度と距離でカリウムの振動がバイオフィルム中を伝播すること
- 細胞外カリウムを一時的に増加させると細胞が脱分極と過分極を順に示すこと(ThTにより確認)
- 過分極中に細胞外カリウム濃度が上昇すること
- 唯一の既知のカリウムチャネルであるYugOがその応答に必要であること
- グルタミン添加が過分極を抑制すること
- グルタミン枯渇がYugO依存的に細胞内カリウム放出を引き起こすこと
をそれぞれ確認しました*3。 これらの実験結果をまとめると、
- 細胞のグルタミン枯渇がカリウムチャネルYugOを通したカリウム放出を促し、
- 放出されたカリウムが周囲の細胞の脱分極と過分極を促してカリウム放出を引き起こし、
- バケツリレー方式にカリウムイオンの波がバイオフィルム中を伝播していく
というストーリーが浮かび上がってきました。実際にHodgkin-Huxleyモデルをアレンジした数理モデルで解析してみたところ、YugOの存在が物理的に離れた細胞同士の電気的情報伝達に十分(Sufficient)だということが示されました。
プロトン駆動力に依存する細胞外グルタミン酸取り込みと細胞内アンモニウム維持の効率が細胞の脱分極によって低下するので、栄養枯渇に陥った細胞がカリウムを放出して周囲の細胞を脱分極させることで成長を停止させ、不足していた栄養の補充を行い、その後の過分極によって栄養取り込み能力を回復するというメカニズムが示唆されました。これが隣の細胞でまた繰り返され、バイオフィルム全体を通して電気信号の伝播と細胞成長速度の協調が行われるのです。
最後に
この論文は近年盛り上がっているQuantitative Biology(Qbio)という分野特有の「定量的に生命システムを説明する」というテーマに取り組んでいて、興味にドハマリしたので選びました。 1月のQbio MeetingでGürol Süelの講演を聞いてから大ファンなのですが、この論文も「変な現象」をビシっとエレガントに説明する、Süelラボらしい名作だと思います。 あと単純に微生物学徒としては微生物が変なことをしていると嬉しいですね。
僕は修士課程の研究で大腸菌の1細胞計測を行っているのですが、微生物の自然界における本来の姿であるバイオフィルム、つまり集団での現象にもとても興味があります。この論文で示されているように、Qbioの強みである「よく定義されたシステムの動態を実験と理論の両面から定量的に解析する」という研究手法は今後生命科学の発展にますます重要な位置を占めていくと思われます。実験と理論、両方できるつよい院生になりたいな、ならなくちゃ、絶対なってやる〜〜〜*4
RPy2を使ってPythonからRを呼び出してデータ解析
Pythonで書いたコードの中でRを呼び出す必要に迫られたので、備忘録を兼ねてやり方を記録しておきます。
この前から取り組んでいるPPiプロジェクトの一環で、タンパク質相互作用ネットワークを超幾何分析してスコアを記録しよう、みたいな話になった。
超幾何分析自体の話はbeyond the scopeすぎるので専門家に譲るとして、分析そのものはphyperというR言語の関数でちゃちゃっとできる。
phyper(q, m, n, k, lower.tail = FALSE, log.p = FALSE)
q:調べる蛋白質のモジュール内のリンク数
m:モジュール内のリンクの総数
n:モジュール外のリンクの総数
k:調べるタンパク質の全リンク数
それで、この前Pythonで生成したファイルにRを走らせるのが一番単純な手法かもしれないけど、面倒だしスマートじゃないので、やはりPythonコードのなかでRを動かしたいところ。
少し調べると、PythonとRの連携はRPy2というパッケージが人気の模様。
ちゃんとPython 3.xでも動作するということで、試してみる。
まずは$ sudo pip install rpy2 みたいな感じでローカルにRPy2をインストール。
それからPythonコードで読み込みます。
from rpy2.robjects import r
Rの関数の引数としてPython側の変数を使いたいものの、どうもうまくいかない。 調べてみると直接使わずにR側の新しい変数にポートして活用するらしい。
r.assign('RVariable', PyVariable)
という感じ。 これを引数に使う変数全部に対して行って、いよいよ関数にぶちこみます。
r('関数名(引数1,引数2,...)')
実際のコードでは、まずnamelist1とwholelistからそれぞれモジュール内のリンクの総数moduleedgesとタンパク質相互採用ネットワーク全体のリンク数wholeedgesを計算し、wholeedgesからmoduleedgesを引くことでモジュール外のリンクの総数nonmoduleedgesを得ました。
それから、モジュール内のタンパク質の名簿であるpnamelistをループで回して、各タンパク質のモジュール内リンク数と総リンク数をそれぞれRにポートし、最後にそれをもとに超幾何分析を行ってdict型に収納、という手順を踏みました。
from rpy2.robjects import r moduleedges = len(namelist1) wholeedges = len(wholelist) nonmoduleedges = wholeedges - moduleedges from rpy2.robjects import r #超幾何分析結果を収納するdictを用意 hypergeo = {} #pythonの変数をRの変数にポート r.assign('module', moduleedges) r.assign('nonmodule', nonmoduleedges) #モジュール内の各タンパクについて超幾何分析 for x in pnamelist: r.assign('intralink', intralink[x]) r.assign('wholelink', wholelink[x]) p = r('phyper(intralink, module, nonmodule, wholelink, lower.tail = FALSE, log.p = FALSE)') #dictに分析結果を収納 hypergeo[x] = p[0]
あとは前回のようにcsvに出力して終わり。
僕以外にも統計分析にはRを使うけどそれ以外は全部Python、みたいな人は多いらしく、今回使ったRPy2のようにPythonの中でネイティブなRを走らせる以外にも、Rの大人気グラフ作成パッケージggplot2をPythonで再現する試みとかいろいろあるみたいです。
やっぱりデータ分析分野はR言語で書かれた遺産が多いので、うまく使っていきたいところです。
今回参考にしたウェブサイト
R: The Hypergeometric Distribution
http://rpy.sourceforge.net/rpy2/doc-2.1/html/
卒論生はビッグデータの夢を見るか
しばらく前に、大規模コホート研究のデータを使って、誕生月と健康状態の相関を調べ、それをもとにめっちゃあたる星座占いをつくったら面白いんじゃないのと思ったわけだけど、肝心のデータがそんなに簡単に入手できそうもない。
コホート研究の論文ってだいたいPublicly available dataを使ってるって書いてあるけど、どこに転がってるんでしょ。
日本人のコホート研究もそれなりに存在するみたいだけど、ちゃんと研究代表に連絡して了承を取らないとデータもらえないような雰囲気。
誕生時期と研究の関係そのものについてはすでに研究がある程度進んでいるようなので、他人の研究に乗っかってもいい気はするけど、自分でデータをこねくり回したい気持ちもある。
あーどっかにコホートデータ転がってないかなァ。